思维导图
本篇文章为解读导师发给我的一篇学姐的论文,目标是能读懂这篇文章中用到的结构和方法,做到读完之后能调通文章的代码。
为什么要研究医学的命名实体识别
医学命名实体是指在医学领域中具有一定特殊含义的词语,例如疾病名称、药物名称、手术名称等。而一般命名实体则是泛指在其他领域中具有一定特殊含义的词语,例如人名、地名、组织机构名等。由于医学领域的专业性和术语多样性,医学命名实体相对于一般命名实体来说更加复杂和难以识别。
例如,有一些疾病名称长且复杂“芳香族L─胺基酸类脱羧基酶缺乏症”,NER网络可能比较难识别出这么长的复合实体。
例如,有一些药物名称经常使用缩写,“阿莫西林”可以简称为“AMX”,并且在不同国家使用的缩写可能还不一样。
因此,在医学领域中进行命名实体识别需要更加精细和专业的处理方式。
目前命名实体识别的主流模型
BiLSTM模型
BiLSTM是RNN网络的一个变体,用于解决序列到序列的问题。在命名实体识别中,BiLSTM其实就是做了一个分类任务。
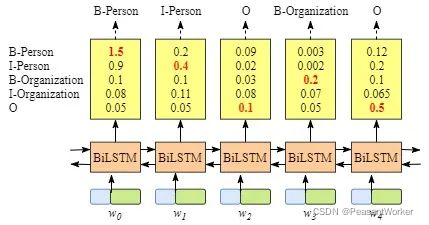
对于输入的每一个字,预测其对应的标签。然后将结果输出,做到命名实体识别的效果。
但单独的BiLSTM命名实体识别效果往往不是很好。
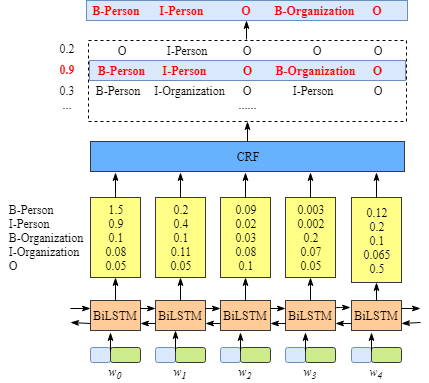
BiLSTM+CRF模型
在BiLSTM后加上一个条件随机场,CRF会将不合理的预测标签修正,并且CRF的解码层会用动态规划的思想找到全局最佳的解法。因此识别正确率会大大增强
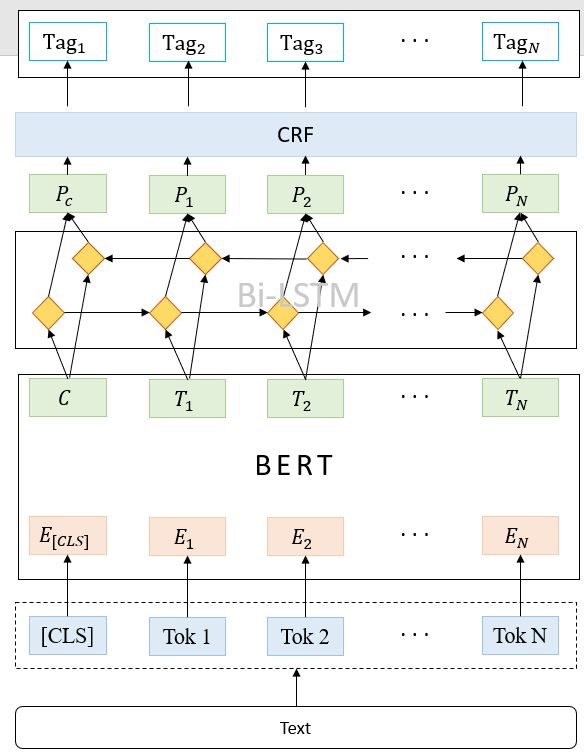
BERT+BiLSTM+CRF模型
这个模型在上一个模型的基础上又加上了预训练层BERT(其实是吧BiLSTM的embedding层替换成了BERT),BERT做的事只有完形填空和判断两个句子是否是连接的。看上去BERT做的事与命名实体识别没什么关系,但最后的效果却实打实的提升了。
本文使用的模型LEBERT+双GUR+star-transform+CRF
首先,为应对专业术语导致的识别难题,文中提出了一种增强边界信息的模型,通过在BERT预训练模型底层融合医学领域词典信息,并引入实体边界预测辅助模块,以提升模型对专有名词的识别能力。其次,针对长实体识别效果不佳的问题,设计了一种双位置编码注意力模型,通过同时考虑字符间的绝对位置和相对位置信息,有效提升了模型对长实体的识别精度。
该模型从隐形和显性两方面增强边界信息:
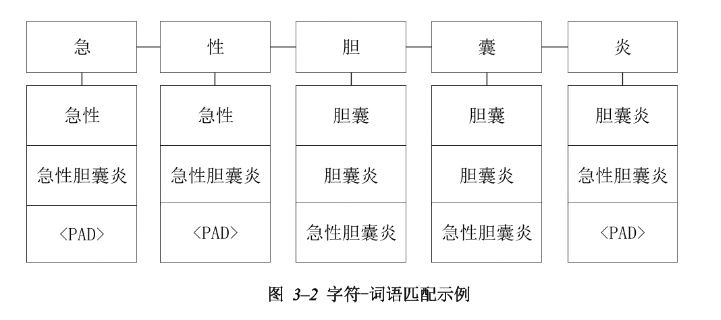
- 一是引入外部医学领域词典信息,增强模型对词语边界的学习能力,隐性增强词汇边界特征;
- 二是增加实体边界预测辅助模块,通过两个双向循环门控单元(GRU)来分别预测实体的头和尾,显性增强实体的边界信息
增强边界信息后实验表明增加了识别的准确率,如在CCKS2020数据集上识别精度F1值相比基线模型提升了2.08%,在CHIP2020数据集上F1值达到了62.98%,在自建乳腺癌超声数据集上F1值达到了97.39%。
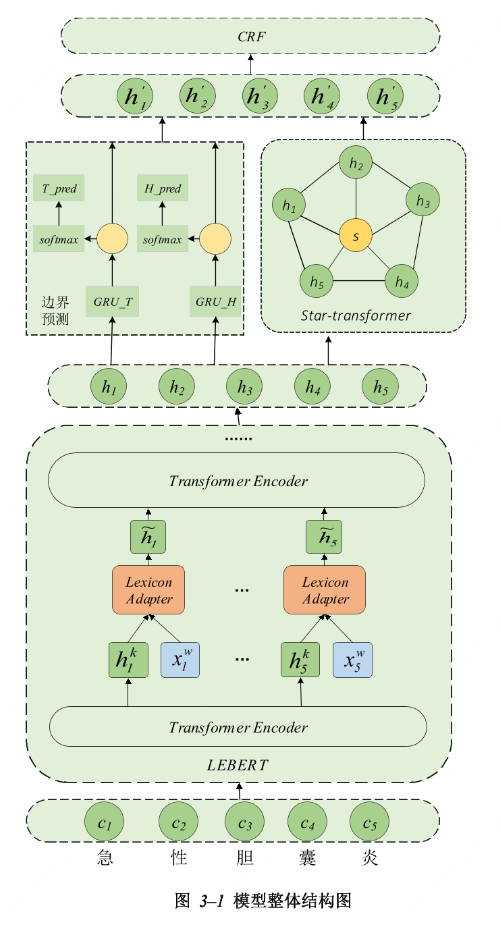
这里用来提取特征做分类的模型是star-transform,由于原transform里的自注意力机制在看一个字的时候需要把其他所有字都看一遍,所以加大了训练的负担。而star-transform模型采用星型结构,只重视相邻的两个字。大大减轻了训练的负担。
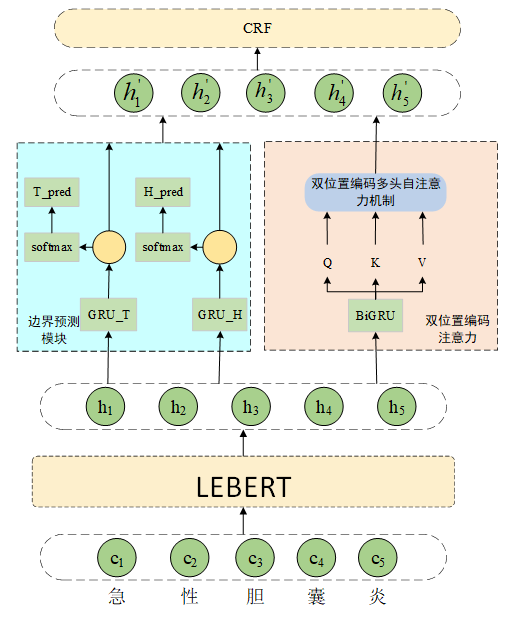
本文改进后的模型LEBERT+双GRU+双位置编码+CRF
star-transform模型虽然有效的减少了训练负担,但由于其只重视相邻的字,导致其对长实体的识别效果不好。因此文章采用双位置编码模型替代了star-transform模型。
双位置编码注意力模型,通过在自注意力计算之前添加绝对位置编码以区分不同位置字符,且在注意力计算过程中引入相对位置编码以获取字符之间的相对位置和方向信息,(绝对位置编码只能区分每个输入的字符,不能得到这些字符间的相对位置关系)从而实现将两种位置编码与输入文本的语义信息进行深度交互,增强上下文特征的提取能力,提升模型识别命名实体的准确率。实验结果表明该模型在CCKS2020数据集上取得的精确率、召回率和F1值都提高了;同时,在长实体占比较大的疾病与诊断实体和手术实体上,相比于基线模型,F1值也分别获得了提升。另外,对于通用领域文本,该模型也有优秀的识别性能,如在Resume数据集的精确率、召回率和F1值也提高了。



Comments NOTHING